Schnelle und sensitive Identifizierung multiresistenter Keime
Forschende der Universität Basel haben ein empfindliches Testsystem entwickelt, mit dem schnell und zuverlässig Resistenzen von Bakterien nachgewiesen werden können. Das System basiert auf winzigen, funktionalisierten Federbalken, die sich bei Bindung von Probenmaterial verbiegen. Bei den Untersuchungen reichte die Probenmenge von nur 1-10 Bakterien aus, um einen Nachweis von Resistenzen zu liefern.
07. Dezember 2020
Bakterien, die sich mit verschiedenen Antibiotika nicht mehr bekämpfen lassen, stellen eine grosse Bedrohung für unsere Gesundheit dar. Medizinerinnen und Mediziner benötigen schnell die Information über bestehende Resistenzen bei einer bakteriellen Infektion, um rasch und richtig reagieren zu können.
Cantilever-Systeme als Alternative
Klassische Methoden für den Nachweis von Resistenzen basieren auf der Kultivierung der Bakterien und der Testung ihrer Empfindlichkeit gegenüber einem Spektrum von Antibiotika. Bis zum Ergebnis verstreichen 48 bis 72 Stunden und manche Bakterienstämme lassen sich zudem nur schlecht kultivieren. Sehr viel schneller funktionieren molekularbiologische Tests, die Resistenz-Gene oder bestimmte kurze Abschnitte im Erbgut mittels PCR (Polymerase-Ketten-Reaktion) vervielfältigen. Auch diese Methode liefert jedoch nicht für jedes Bakterium befriedigende Ergebnisse.
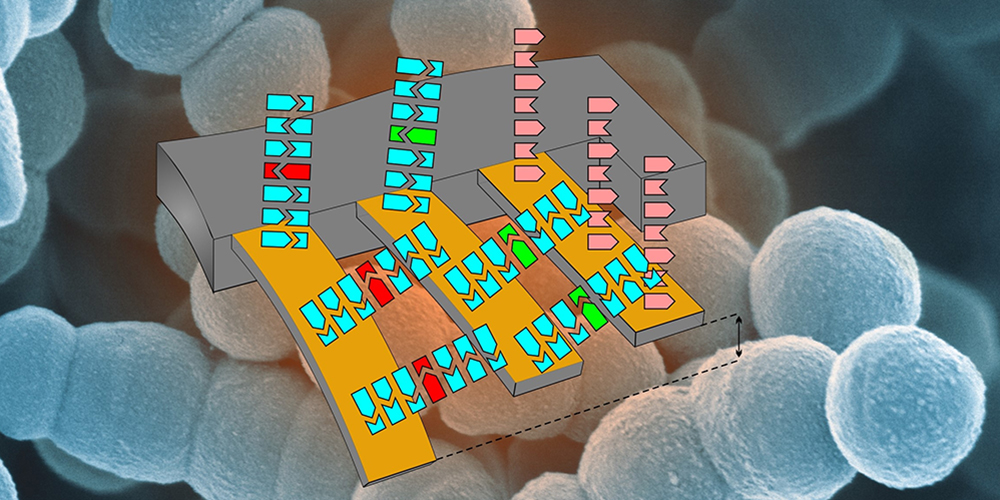
Eine Alternative bieten Methoden, die winzige Federbalken (Cantilever) einsetzen. Diese Federbalken verbiegen sich nachweisbar, wenn beispielsweise auf ihrer Oberfläche RNA-Moleküle binden. RNA-Moleküle sind «Arbeitsabschriften» der Gene und dienen unter anderem als Anleitung zum Bau von Proteinen. Über die RNA-Moleküle lassen sich auch Resistenzgene im Erbgut der Bakterien nachweisen.
Ohne Markierung und Vervielfältigung
Ein Team von Wissenschaftlern des Departements Physik, des Departements Biomedizin und des Swiss Nanoscience Institutes der Universität Basel stellt nun im Fachjournal «Global Challenges» ein Cantilever-Testsystem vor, mit dem sie RNA von einzelnen Bakterien nachweisen konnten, die auf Resistenzen gegenüber Antibiotika schliessen lassen. Die Proben für die Analyse müssen bei dem neuen Cantilever-System weder vervielfältig noch markiert werden.
Zunächst fixierten die Forschenden dazu Abschnitte dreier Gene, die mit der Resistenz gegenüber Vancomycin assoziiert sind, auf den Cantilevern. Anschliessend liessen sie einen RNA-Extrakt aus Bakterien über die so vorbereiteten Federbalken fliessen. Waren RNA-Moleküle der Resistenzgene vorhanden, kam es zur Bindung der übereinstimmenden RNA-Stücke auf den vorbereiteten Cantilevern. Diese Bindung führt zu einer Auslenkung der Federbalken im Nanometerbereich, die sich mithilfe eines Lasers detektieren lässt.
Eindeutiges Signal auch bei Punktmutationen
Es liessen sich mit dieser Methode nicht nur Resistenzgene nachweisen, sondern auch einzelne Punktmutationen, die mit Resistenzgenen assoziiert sind. Für diese Untersuchung verwendeten die Forschenden Punktmutationen, die mit Genen gekoppelt sind, die für Resistenzen gegen Ampicillin und andere Betalaktam-Antibiotika verantwortlich sind.
«Der grosse Vorteil der von uns entwickelten Methode ist ihre Schnelligkeit und Sensitivität», sagt Dr. François Huber, Erstautor der Arbeit. «Wir konnten innerhalb von fünf Minuten winzige Mengen von spezifischen RNA-Stücken nachweisen.» Die nachgewiesenen Mengen, wenn es um einzelne Mutationen ging, entsprachen etwa 10 Bakterien. Beim Nachweis ganzer Resistenz-Gene erhielten die Forschenden bereits bei einer RNA-Menge, die einem einzelnen Bakterium entspricht, ein eindeutiges Signal.
«Wenn wir bestimmte Gene oder Mutationen im Genom der Bakterien nachweisen können, wissen wir, welche Antibiotika-Resistenzen die Bakterien aufweisen», erklärt Prof. Dr. Adrian Egli vom Universitätsspital Basel, dessen Team massgeblich an der Studie beteiligt war.
«Für unsere Arbeit im Spital wäre es wünschenswert, derartig schnell zuverlässige und sensitive Informationen über die Resistenzen von Keimen zu bekommen.»
Originalpublikation
François Huber, Hans Peter Lang, Daniela Lang, Daniel Wüthrich, Vladimira Hinić, Christoph Gerber, Adrian Egli, Ernst Meyer
Rapid and ultrasensitive detection of mutations and genes relevant to antimicrobial resistance in bacteria
Global Challenges (2020), doi: 10.1002/gch2.202000066
Weitere Auskünfte
Prof. Dr. Ernst Meyer, Universität Basel, Departement Physik, Tel. +41 61 207 37 24, E-Mail: ernst.meyer@unibas.ch
Dr. François Huber, Universität Basel, Departement Physik, Tel. +41 61 207 37 24, E-Mail: francois.huber@unibas.ch
Prof. Dr. Christoph Gerber, Universität Basel, SNI Departement Physik, E-Mail: christoph.gerber@unibas.ch